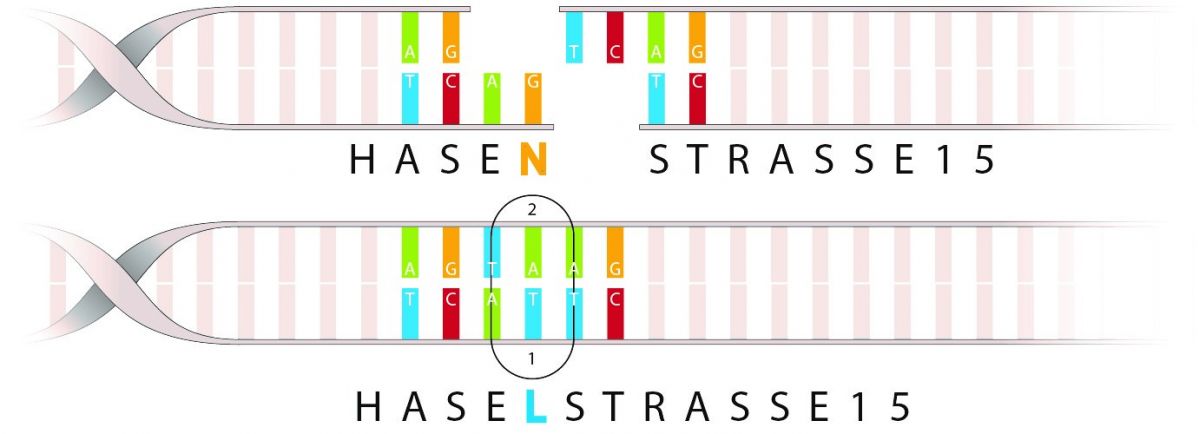
Wie kann so etwas passieren? Stellen Sie sich vor, Sie laden ihre Freunde zu einer Party ein und geben die Adresse, zum Beispiel Hasenstraße 15, telefonisch durch. Die meisten der Partygäste werden die Partylocation finden, aber ein paar Gäste werden die Adresse falsch verstanden haben und zum Beispiel bei der Haselstraße 15 klingeln. Ähnlich ist das bei der Navigationseinheit der Genome Editing Methoden. In der Regel suchen die Forschenden für ihre Navigationseinheit eine Sequenz aus, die nur einmal im ganzen Genom vorkommt. Manchmal verbindet sich die Navigationseinheit jedoch mit einer DNA-Sequenz, die der Zielsequenz sehr ähnlich ist und sich nur in einzelnen Basen unterscheidet. Verhindern können die Forschenden das, indem sie als Zielsequenz eine Basenfolge wählen, die sehr selten vorkommt oder sehr lang ist. Also nicht gerade die Hasenstraße, die leicht zu Missverständnissen führt.
In der Übersichtsstudie „What is the available evidence for the range of applications of genome-editing as a new tool for plant trait modification and the potential occurrence of associated off-target effects: A systematic map“ haben Forschende des JKI alle englisch- und deutschsprachigen Veröffentlichungen zu genomeditierten Pflanzen, die es zwischen Januar 1996 und Mai 2018 gab, zusammengetragen. Sie haben unter anderem das Auftreten von off-target Effekten und deren Messmethoden analysiert. Sie fanden 252 Experimente, bei denen Off-Target Effekte untersucht wurden.
Wie identifizieren Forschende die off-target Effekte?
Nehmen wir an, das Genom einer Pflanze entspricht einer Stadt und die DNA-Sequenzen einzelnen Adressen. Als Zieladresse haben die Forschenden die Hasenstraße 15 ausgewählt. Es gibt zwei Ansätze, um verirrte Gäste bzw. off-target Effekte zu finden. Entweder suchen die Forschenden das gesamte Genom nach off-target Effekten ab (unbiased detection Methode) oder sie suchen gezielt an potentiellen Orten, die eine hohe Ähnlichkeit mit der Zieladresse haben (biased detection Methode).
Wenn es sich um kleine Genome handelt, kann man sich vorstellen, dass eine Analyse des gesamten Genoms eine gute Möglichkeit ist, um off-target Effekte zu identifizieren. Große Genome, wie zum Beispiel das Weizengenom, ähneln dagegen den Ausmaßen einer Milliardenmetropole. In diesem Fall kann die Überprüfung aller Straßenzüge sehr aufwendig werden. Hier kann es sinnvoller sein, die Suche auf potentielle Fehladressen (off-targets) einzugrenzen.
Im letzteren Fall (biased detection Methode) überlegen die Forschenden wie viele potentielle Möglichkeiten es gibt, die Adresse falsch zu verstehen. Sie wählen zum Beispiel die Anzahl der Buchstaben aus, von denen sie erwarten, dass sie von den Gästen falsch verstanden werden können. Das wäre dann so, als ob die Gastgeber im Telefonbuch nach Adressen suchen, die der Zieladresse (Hasenstraße 15) sehr ähneln. Um beim Beispiel Hasenstraße zu bleiben, wären mögliche Veränderungen mit einem Buchstaben Haselstraße, Basenstraße, Hase-straße, Haasenstraße oder Vasenstraße. Veränderungen mit zwei Buchstaben wäre zum Beispiel Baselstraße oder Paasenstraße usw. Der Nachteil an der Methode ist, dass der Gastgeber nicht sicher sein kann, dass einzelne Partygäste nicht doch bei einer Adresse klingeln, die er nicht berücksichtigt hat.
Dann erst beginnt die tatsächliche Suche nach den verirrten Gästen beziehungsweise den off-target Effekten. Wie mit einem Fernglas schauen sich die Forschenden die DNA an, indem sie die DNA mehrfach kopieren und dann auf Veränderungen im Genom im Vergleich zum Genom der Ausgangspflanze untersuchen.
Die Ergebnisse der Übersichtsstudie zeigen, dass die am häufigsten verwendete Genome Editing Methode, mit über 90%, CRISPR/Cas ist. Hier kam es im Durchschnitt an etwa 3% von den mittels der biased detection Methode identifizierten potentiell falschen Adressen tatsächlich zu off-target Effekten. Das heißt also, dass bei ungefähr drei von hundert potentiell falschen Adressen Partygäste klingeln. Bei der Sequenzierung des gesamten Genoms, einer unbiased detection Methode, wurden keine off-target Effekte gefunden, die der CRISPR/Cas Navigationseinheit zugeordnet werden konnten. Die Befunde der anderen Genome Editing Methoden TALENs, ZFN, Meganukleasen und ODM bewegten sich zwischen 0 und 3%. Diese Zahlen sind jedoch wenig aussagekräftig, da es gerade in der Grundlagenforschung viele Studien gibt, in denen absichtlich Sequenzen ausgewählt werden, die weitere ganz ähnliche Sequenzen im Genom haben. Dies wird gemacht, um neue Erkenntnisse über das Auftreten von off-target Effekten zu gewinnen.
Folgen die Forschenden der Empfehlung die Zielsequenz so zu wählen, dass keine weiteren ähnlichen DNA-Sequenzen vorhanden sind, sinken die off-target Effekte unter 1%.
Die Autoren dieser Veröffentlichung betonen daher, dass sie mit dieser Analyse noch keinen abschließenden Rückschluss auf das generelle Auftreten von off-target Effekten zielen können, da die Methoden und Annahmen, die die Forschenden in den Studien nutzen sehr heterogen sind. Außerdem lassen die geringe Anzahl der Experimente, wie beispielsweise bei der unbiased detection Methode, keine valide Aussage zu. Hier ist eine tiefergehende Analyse nötig.
Die Studie endet nicht mit dieser Veröffentlichung, sondern wird noch bis Ende 2019 fortgesetzt. Es ist zu erwarten, dass in Zukunft die Navigationseinheiten der einzelnen Genome Editing Methoden noch weiter verbessert werden.
Zusatzinfo:
Welche Bedeutung haben off-target Mutationen im Kontext der Pflanzenzüchtung?
Die Veränderung der DNA durch Mutationen, ist die Grundlage der Anpassung der Lebewesen an seine Umwelt. Untersuchungen zeigen, dass die spontane und natürliche Veränderung des Genoms von einer Generation zur nächsten im Modellorganismus Arabidopsis thaliana bei einer Mutation pro 150 Millionen Basenpaaren liegt. Beim Weizengenom, mit seinen 15 Milliarden Basenpaare, liegt diese bei bis zu 200 Veränderungen. Diese natürliche Mutationsrate wird in der Pflanzenzüchtung schon seit vielen Jahrzehnten durch den Einsatz von Strahlung oder Chemikalien je nach Dosis um den Faktor 700 künstlich erhöht. Sorten, die mit solchen Methoden entstanden sind, finden sich heute in vielen unserer im Supermarkt erhältlichen Nahrungsmittel (egal ob biologisch oder konventionell). Mutationen, die spontan oder durch den Einsatz von Chemikalien oder Strahlung entstehen unterscheiden sich nicht von on-target und off-target Mutationen, die durch die Verwendung von Genome Editing entstanden sind.
Mutationen, die keinen mess- oder sichtbaren Einfluss auf die Qualität und die Eigenschaften der Pflanzen haben, werden in der konventionellen Pflanzenzüchtung vernachlässigt. Pflanzen, die unerwünschte Eigenschaften zeigen, die durch ungewollte Mutationen (off-target Effekte) egal durch welche Technik hervorgerufen wurden, werden entweder von den Pflanzenzüchtern verworfen oder sie entfernen die Mutationen mittels Rückkreuzung aus dem Genom. Daher spielen off-target Effekte in der Pflanzenzüchtung kaum eine Rolle.
Quellenangaben
Dominik Modrzejewski et al. (2019) Systematic Map-Protocol: What is the available evidence for the application of genome editing as a new tool for plant trait modification and the potential occurrence of associated off-target effects? A Systematic Map. Environmental Evidence 8:27 https://environmentalevidencejournal.biomedcentral.com/articles/10.1186/s13750-019-0171-5