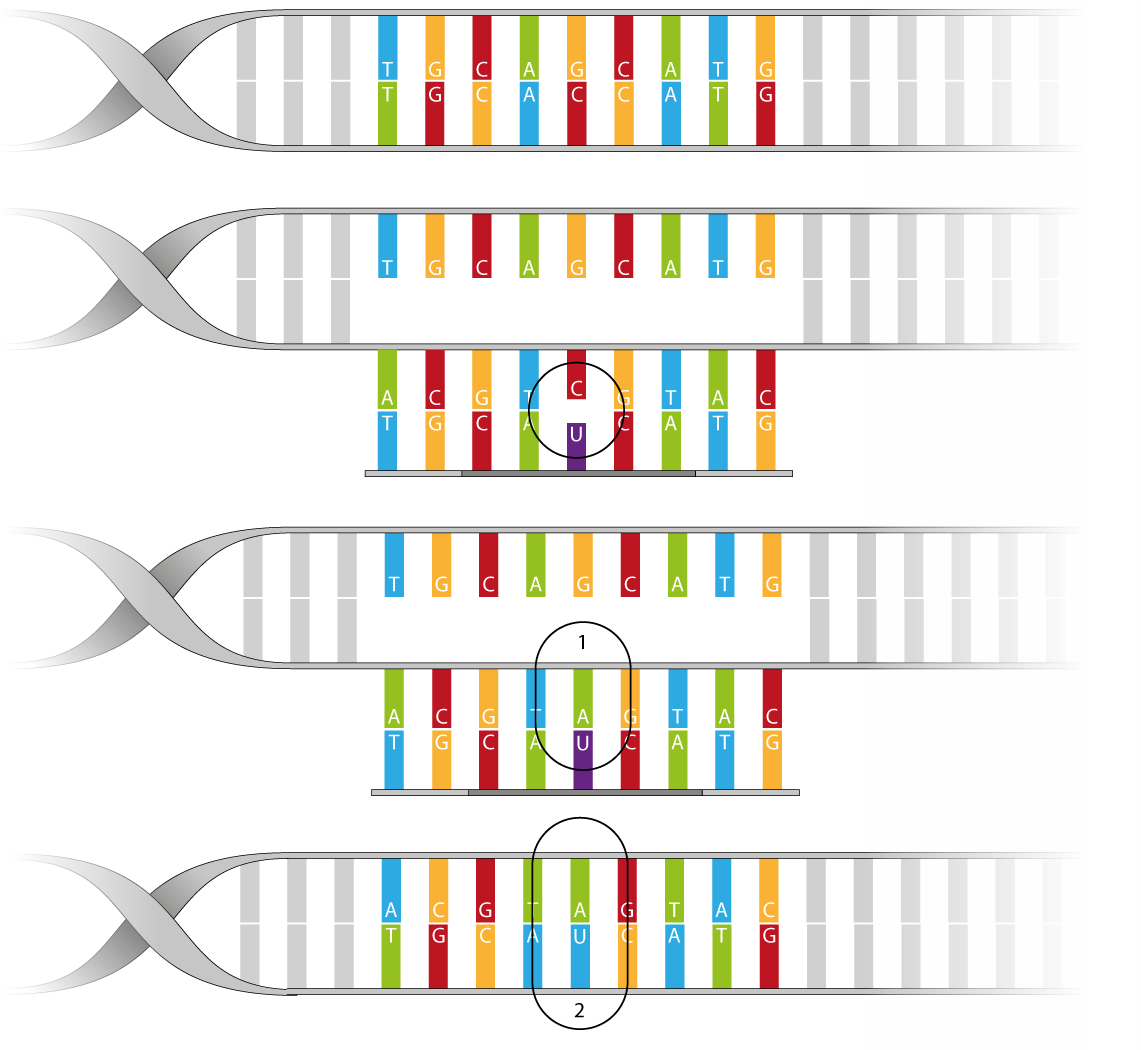
Bei der Oligonukleotid gerichteten Mutagenese wird ein kurzer veränderter DNA-Abschnitt genutzt, welcher sich an einer oder wenigen Stellen von der Zielsequenz unterscheidet, um an einer definierten Stelle eine Mutation im Erbgut zu induzieren (Punktmutation).
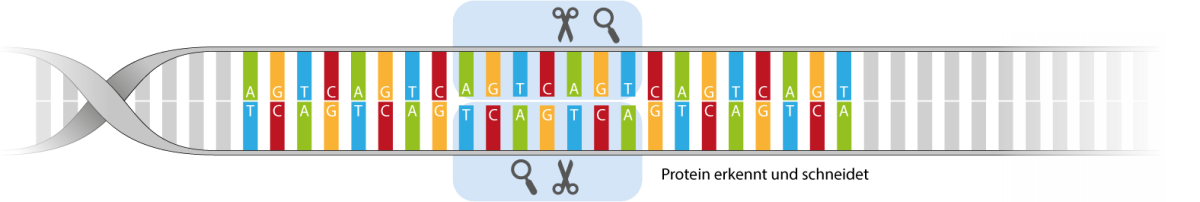
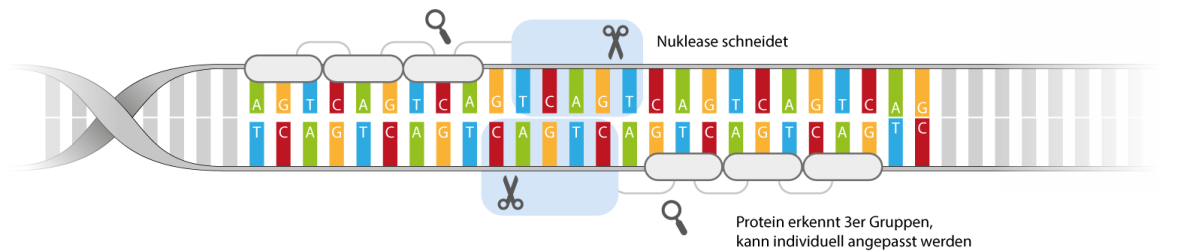
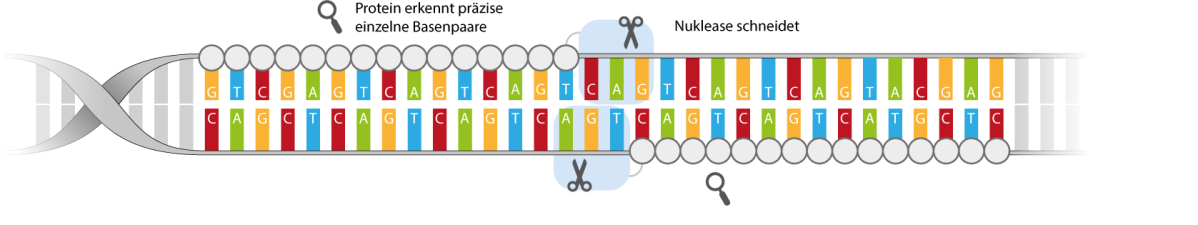
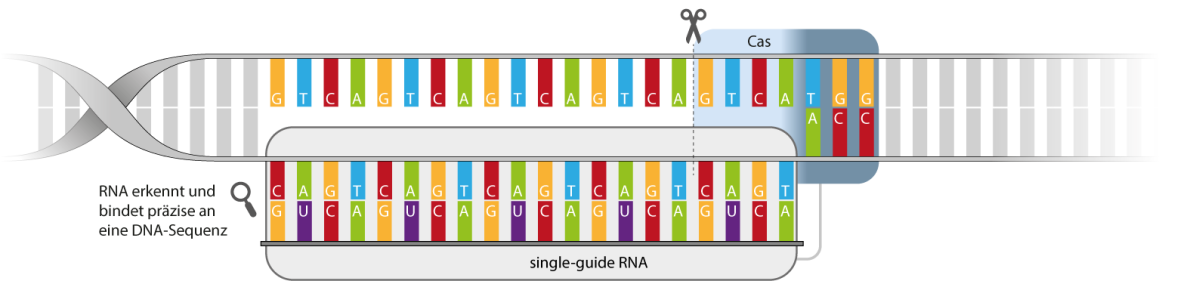
Die ortsspezifischen Nukleasen bestehen aus zwei Elementen. Ein Element des Systems erkennt die zu verändernde Erbgutsequenz, ein zweites schneidet das Erbgut an eben dieser Stelle. Es gibt vier sich ähnelnde Systeme ortsspezifischer Nukleasen: die Meganukleasen, die Zink-Finger-Nukleasen (ZFN), Transcription Activator Like Effector Nukleasen (TALEN) und das Clustered Regularly Interspaced Short Palindromic Repeats/CRISPR associated Systems (CRISPR/Cas System).
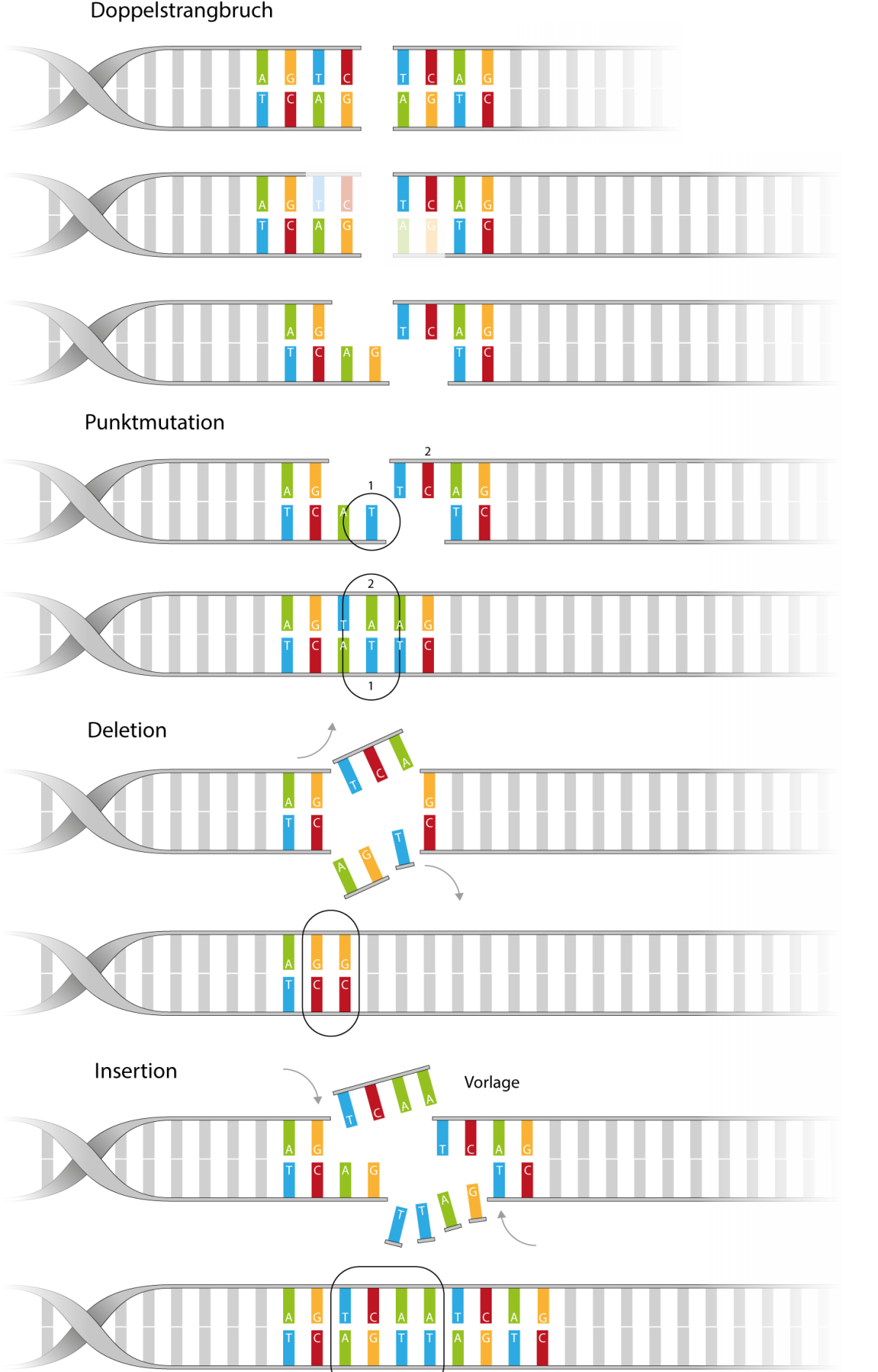
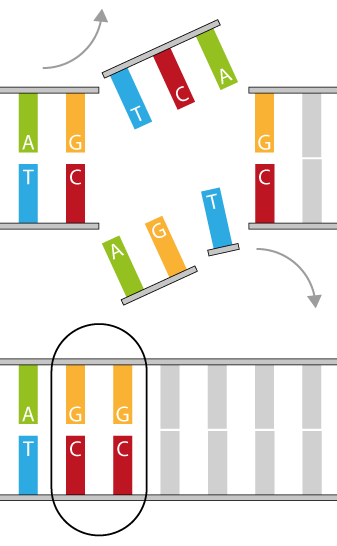
Die Reparatur der durch die neuen Techniken erzeugten Brüche orientiert sich dabei an den natürlichen molekularen Reparaturmechanismen der Zelle, die zum Beispiel Fehler beim Kopieren von Erbgut in den Zellen beheben. So können einzelne Bausteine des Erbguts ausgeschnitten oder neu kombiniert werden. Bedingt durch die zelleigenen Systeme können so auch einzelne Bausteine hinzufügt werden, ähnlich wie bei natürlichen oder induzierten Mutationen durch Chemikalien oder Bestrahlung. Allerdings besteht ein gravierender Unterschied darin, dass die Veränderungen nicht wie bei Mutationszucht ungerichtet erfolgen, sondern gezielt an gewollten Stellen.
Die Funktion des Genome Editing lässt sich in drei Schritten beschreiben:
- Es werden Lotsen (guides) genutzt, wie DNA/RNA-Abschnitte oder Proteine, welche die zu verändernde Erbgutsequenz erkennen.
- Die zu verändernde Sequenz wird von speziellen Proteinen, den ortsspezifischen Nukleasen, geschnitten (nur bei ortsspezifischen Nukleasen).
- Der Schnitt oder die Fehlpaarung wird durch natürliche zelleigene Systeme repariert.